3가지 과일 이미지를 Clustering으로 모아봅니다.
비지도 학습에 속하는 K-means clustering과 PCA에 대해 정리하는 글입니다.
K-means clustering
데이터를 K개의 군집으로 모으기 위한 알고리즘입니다.
과정

- 무작위로 k개의 centroid (클러스터 중심)를 정합니다.
- 각 샘플에서 가장 가까운 cluster center를 찾아서 해당 cluster의 샘플로 지정합니다.
- cluster에 속한 샘플의 평균값으로 centriod를 변경합니다.
- centroid에 변화가 없을 때 까지 (수렴할때 까지) 2번부터 반복합니다.
실습
데이터 준비
데이터를 다운받고 읽습니다. npy 포맷이므로 numpy로 읽습니다.
1 | # 다운로드 |
데이터 구조를 확인합니다.
1 | fruits.shape |
데이터셋은 3차원이고, 300개의 과일이 100x100의 픽셀로 된 이미지입니다. 과일의 모양새는 아래와 같습니다.
과일 종류: 사과, 파인애플, 바나나
과일들은 3가지로 되어있습니다. 데이터 순서대로 100개씩 총 300개입니다. 여기서 유사한 과일끼리 묶어보려 합니다.
tabular data와 달리 차원이 존재합니다. 2차원 이미지 형태의 데이터간에 유사함을 파악하여 비교하려면 어떻게 해야할까요?
픽셀 단위로 쭉 나열한 하나의 배열로 만들면 됩니다. numpy의 reshape 메소드를 이용해보겠습니다.
1 | fruits_2d = fruits.reshape(-1, 100*100) |
reshape는 사용자가 원하는 형태의 array로 만들 수 있습니다.parameter에는 1차원 개수, 2차원 개수, ... 가 입력됩니다. 데이터셋은 300개의 샘플, 너비 100픽셀, 높이 100픽셀인 3차원 이었고, 이를 2차원으로 만들기 위해 2차원에는 100100 (너비높이)를 입력하였고 1차원에는 -1을 넣었습니다. -1은 알아서 맞춰줍니다.
KMeans로 모델을 만들어보겠습니다.
1 | from sklearn.cluster import KMeans |
각 샘플별로 어떻게 찾았는지 확인해보겠습니다. labels_에는 각 샘플의 레이블이 나타납니다.
1 | print(km.labels_) |
개수별로 카운트해봤을 때에도, 100개씩 묶어주긴 한거 같습니다. 어떻게 나왔는지 그림으로 살펴보겠습니다.
라벨 0

라벨 1

라벨 2

과일별로 대체로 잘 모여있는것 같군요.
각 라벨의 centroid는 km.cluster_centers_에 2차원으로 존재합니다. 이를 3차원으로 바꾸고 출력하면 다음과 같습니다.
각 라벨의 centroid 출력
여러가지 회전한 형태의 각각의 과일을 학습해서 평균점을 찾다보니 흐릿하게 뭉개진것 같습니다. 그래도 3가지를 확실히 구분할것 같긴 하네요.
여기서는 K를 3으로 지정해주었습니다. 하지만 실제 데이터에서는 몇 가지의 과일이 있는지 모릅니다. 그러면 적당한 cluster 개수 K는 어떻게 알아낼까요?
최적의 K 값은 알아내기 어렵습니다. (예전에 지도교수님은 이걸로 책 몇권을 낼 수 있다고도 하신 기억이 나네요)
Elbow
k means clustering은 centroid와 cluster에 속한 샘플 사이의 거리를 계산할 수 있습니다. 이 거리의 제곱을 하여 합진 값을 inertia라고 하고, 데이터들이 가깝게 모인 정도를 나타냅니다.
scikit-learn의 KMeans 모델로 clustering을 진행하면 intertia_도 같이 저정됩니다.
실습한 과일 데이터로 K의 개수별로 어떤일이 일어날지 상상해 봅시다…
- k=1: 3가지 과일을 1개의 cluster를 찾게 한다면, 데이터의 중심이 될것입니다. 따라서 intertia는 최대치로 높게 나타날 것입니다.
- k=2: 3가지 과일 중 2가지를 clustering을 시킨다면, k=1보다는 아니지만 데이터 중 일부는 여전히 centriod와 거리가 멀으므로 inertia가 높게 나타날 것입니다.
- k=3: 3가지 과일 중 3가지를 clustering하여 이상적인 cluster를 형성한다면 inertia가 급격히 줄어들었을 것입니다.
- k=4~ : 3가지 과일에서 4개 이상의 cluster를 만든다면, 작은 cluster가 생길 것이고, k가 높아질수록 점차 inertia가 낮아 질 것입니다.
따라서, 급격히 내려간 지점 (k=2 -> 3)과 완만히 내려간 지점 (k=3 -> 4)를 찾아내야합니다. Elbow 방법으로 이를 확인할 수 있습니다.
확인을 위해 K가 2~7개 일때, inertia의 변화를 보겠습니다.
1 | # K = 2 ~ 6 으로 clustering 후 intertia를 리스트에 저장 |
기울기를 볼때 k=3에서 변화가 보이시나요?
저 부분을 팔꿈치 같다고 하여 elbow 지점이라 합니다. 이 데이터는 큰 변화는 아니지만 elbow 지점보다 많은 clustering을 시킨다면 inertia가 계속 줄어들게 됩니다.
Principle Component Analysis PCA
PCA는 여러개의 데이터 feature에서 패턴을 알려주고, 어떤 feature가 중요한지와, 해당 feature가 얼마나 데이터를 잘 설명해 주는지 알수 있습니다.
PCA에 대한 이론적인 부분은 제가 좋아하는 유튜브 채널의 영상을 올려드립니다. (한글 자막도 있어요)
실습
scikit-learn에서는 PCA 클래스를 제공합니다.
1 | from sklearn.decomposition import PCA |
n_components를 50으로 지정하였습니다. 주성분Principal component 벡터를 50개 찾는 것입니다. 찾은 값은 components_에 들어있습니다. 구조를 확인해보겠습니다.
1 | pca.components_.shape |
50개의 주성분과 10000개의 feature (100x100)가 들어있습니다.
그림으로 표현하면 아래와 같습니다.

왼쪽 위에서 오른쪽으로 순서대로, 원본 데이터에서 데이터를 잘 표현하는 벡터입니다.
이것이 의미하는 것은 100x100개의 차원이었던 feature에서 가장 잘 설명할 수 있는 50개를 선별한 것입니다. 이제 원본데이터를 주성분 벡터에 투영하면 특성의 개수를 100x100개에서 50개로 줄일 수 있습니다.
1 | fruits_pca = pca.transform(fruits_2d) |
10000개 였던 원본 이미지를 50개로 줄였습니다. 이 50개의 feature를 추정하여 복원하는 기능도 있습니다.
1 | fruits_inverse = pca.inverse_transform(fruits_pca) |
복원된 차원의 벡터로 그림으로 그려보면 다음과 같습니다.
조금 흐릿해지고 배경도 어두워졌지만, 확실히 각각의 과일을 복원시켰습니다. 차원을 줄이고, 복원하는걸 보니 주성분 벡터들이 확실히 잘 설명하는 거 같습니다만 아직은 어떤 주성분 벡터가 얼마나 잘 설명하는지 모르겠습니다.
설명된 분산
주성분이 원본 데이터를 잘 나타내는지 기록한 값을 설명된 분산explained variance이라 합니다.
scikit-learn의 PCA 모델의 explained_variance_ratio_에 50개 주성분에 대한 분산이 저장되어 있습니다. 모두 더하면 원본 데이터에 대한 분산 비율을 얻을 수 있습니다.
1 | exp_var_ratio = pd.DataFrame(pca.explained_variance_ratio_) |
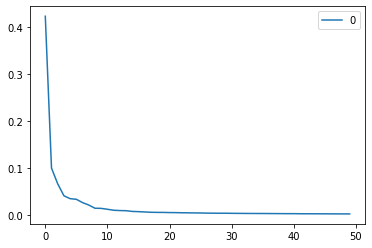
50개의 주성분으로 원본데이터의 92%의 분산을 표현할 수 있습니다.
게다가 첫 번째 주성분 (PC1)으로 데이터의 40% 이상을 표현할 수 있습니다.
이번에는 설명된 분산의 50%에 달하는 주성분을 PCA로 찾아보겠습니다. n_components에 정수가 아닌 실수로 0.5를 입력하면 됩니다.
1 | pca = PCA(n_components=0.5) |
찾아진 주성분은 2개입니다. 위의 그래프를 보시면 아시겠지만 PC1, PC2으로 50% 이상을 표현할 수 있고, 이번에는 n_components에 0.5를 입력하였으므로 PC1, PC2를 찾아준 것입니다.
같은 방식으로 transform을 이용해 원본 데이터를 2개의 차원으로 축소 시켰습니다.
이번에는 LogisticRegression으로 분류를 해보겠습니다.
1 | from sklearn.linear_model import LogisticRegression |
PC1, PC2만으로도 0.99의 정확도를 내었습니다.
이번엔 PC1, PC2으로 K-means clustering을 해보겠습니다.
1 | km = KMeans(n_clusters = 3, random_state = 42) |
원본데이터 전체의 특성을 이용했을 때와 거의 같은 결과입니다.
이번에는 산점도를 그려서 cluster를 어떻게 형성했는지 보겠습니다.
데이터들이 잘 뭉쳐져 있는것이 확인되는군요.
사과와 파인애플은 cluster가 가까이 있음을 확인할 수 있고, 혼동스러운 부분이 있겠습니다. 이렇게 시각화를 해서 데이터를 관찰하면 데이터 분석의 방향성을 고려할 수 있습니다.
여기서 중간점검을 해보겠습니다.
어떤 데이터에서 scikit-learn의 PCA를 이용해 주성분을 10개를 얻었을 때, 설명된 분산이 가장 큰 주성분은 몇 번째일까요?
1️⃣첫 번째 주성분
2️⃣다섯 번째 주성분
3️⃣열 번째 주성분
4️⃣알 수 없음
…
정답은 1️⃣첫 번째 주성분입니다. 찾아진 주성분은 설명된 분산이 큰 순서부터 찾고 나열합니다. 그래서 대부분 PCA를 이용할 때, 첫 번째 주성분 (PC1)과 두 번째 주성분 (PC2)을 결과로 제시합니다.
오늘은 K-means clustering, PCA에 대해 정리해보았습니다.
어느덧 책 한권의 마무리가 되어가네요.
다음에는 인공신경망에 대해 정리하겠습니다.
읽어주셔서 감사드립니다 👋



